-
Genome circular map 그리기genome data analysis/Genome analysis 2020. 4. 1. 14:15
Circular map 그리기
저는 사람 유전체 분석 쪽을 주로 진행했었는데 어느 날... 세균 circular genome map을 그려달라는 부탁을 받았습니다. 그러고 나서 여러 프로그램들을 찾아봤는데 어떻게 사용하는 건지 잘 이해가 안 갔습니다. 아마 2주쯤? 시간을 투자하면 매뉴얼을 읽고 차츰차츰 터득해나갈 수 있었겠지만 이거 그리는데 2주를 다 쓸 수는 없는 상황이었습니다.... 그러고 보니 결국 한 달 정도를 circular map 그리는데 쓴 거 같긴 하네요.. Circular map을 그리는데 BRIG이라는 프로그램을 사용했습니다. 이게 얼마나 사용되고, 얼마나 좋은 프로그램인지는 모르겠습니다만.. 제가 유일하게 사용할 수 있는 circular map 그리는 프로그램입니다. 제가 매뉴얼을 읽고 이해해 나갔다면 오래 걸렸겠지만 다른 박사님 분에게 사용법을 가르쳐달라고 했고 1시간? 가량 그분의 시간을 빌려 어떻게 사용하는지 익혔습니다. 대부분의 유전체 분야 연구자들은 선배들로부터 해당 프로그램들을 배울 것 같은데, 저 같은 경우는 혼자서 오류를 해결하고 프로그램들을 익혀야 하는 경우가 많았습니다. 아마.. 저 같은 사람들이 또 있을 것 같고, 그분들의 시간을 조금이나마 아끼고 다른 데 투자할 수 있게 했으면 하는 마음으로 이 글을 적어봅니다.이 프로그램은 window에서 돌아갑니다. 하지만 아나콘다 사용법을 조금 알아야 합니다. 아나콘다를 깔고 실행하는 방법은 구글링을 하면 많이 찾을 수 있습니다. 여기에서 필요한 과정이 포함된 글은 여기에 포함되어 있네요.
1. BRIG 프로그램을 다운로드합니다. http://brig.sourceforge.net/
BLAST Ring Image Generator (BRIG)
BRIG is a free cross-platform (Windows/Mac/Unix) application that can display circular comparisons between a large number of genomes, with a focus on handling genome assembly data. The application is available at: http://sourceforge.net/projects/brig If yo
brig.sourceforge.net
2. NCBI blast 다운로드(이건 BRIG 매뉴얼 installlation 부분 참고해서 다운로드하면 됩니다. 이까지 매뉴얼을 읽고 실행하는 것은 어렵지 않을 거예요) 합니다. 매뉴얼 안 보실 분들은 여기
http://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
에서 다운로드하면 되는데 주소는 계속 변경될 수 있어요!! 매뉴얼에 있는 주소와도 살짝 다릅니다.
3. BRIG 압축파일 압축을 풉니다.
4. 아나콘다를 실행합니다.
5. 해당 디렉터리(폴더)로 이동해
java -jar BRIG.jar를 타이핑합니다. java가 실행이 안 될 수 있어요. 그건 그때 어떤 오류가 뜨는지 확인하고 구글링을 해봐야 합니다. 제 컴퓨터에는 그냥 실행이 됐네요. 아나콘다 사용법이나 디렉터리를 변경하고 이런 거 역시 잘 모르신다면 먼저 인터넷에서 검색해 찾아보세요.

*빨간 줄: 잘 못 타이핑한 것.
6. Reference file 준비
- reference sequence file은 fna (fasta도 되는지는 안 해봤어요)와 nhr, nin, nsq 파일이 필요합니다.
- fna파일은 얻기 쉽지만 nhr 파일 등은 어떻게 만드느냐... 이 파일들은 명령 프롬프트에
makeblastdb -in [reference genome; fasta] -dbtype nucl -out [output name]치면 됩니다. 대괄호까지 치는 건 아니고 첫 번째 대괄호 부분은 reference genome file 이름을 적으라는 것이고 두 번째 대괄호 부분은 출력되는 nhr, nin 등의 파일 앞에 붙을 이름을 적어주시면 돼요. 해보시면 무슨 말인지 아실 거예요. 문제는.. 이 명령어는 제 컴퓨터에서도 실행이 안 됐습니다. 저는 리눅스 서버에서 해결했지만 윈도 환경에서는 어떻게 해야하는지는 링크해드릴게요.
* 중요한 것은 BRIG 프로그램에서는 ref.fna, ref.nhr, ref.nin 이런 식으로 파일 만들면 circular map 제작이 안 됩니다. ref.fna, ref.fna.nhr, ref.fna.nin과 같은 방식으로 파일 이름을 수정해줘야 합니다.

* 이런 식으로 만드시면 됩니다.
7. Input file 준비
메뉴얼에는 fasta 파일이 된다고 되어있는 거 같은데 실제로는 gbk 파일로만 했을 때 circular map 만드는 걸 성공했습니다.... 영어가 딸려서... gbk 파일은 gene runner http://www.generunner.net프로그램을 다운로드하고 설치한 후 열기 해서 input 염기서열 파일을 넣고 다른 이름으로 저장할 때 input1. gbk와 같은 형식으로 저장하면 됩니다.Gene Runner
www.generunner.net
gbk 파일 만들기
Genome 데이터 분석할 때 gbk 파일이 필요한 경우가 있습니다. 간단하게 DNA sequence만 필요한 경우 아래와 같이 진행하면 됩니다. 아래는 gene runner 프로그램을 이용해 gbk 파일을 만들었습니다. 전체적인 과..
varamos.tistory.com

- 빨간색 화살표의 Preferences > BRIG option을 클릭

- Browse를 클릭. ncbi blast를 다운로드한 위치로 설정해주세요(다운로드 된 폴더를 더블 클릭 후 bin 폴더 한 번만 클릭).

"bin"을 더블클릭하지마시고 한번 클릭 후 열기 버튼 누르기 - 열기 버튼을 누름 > Save & Close 버튼 누름
9. Reference, Input 파일 입력하기
- 각각에 reference와 input 파일(gbk 형식) 을 넣어 주고 output folder를 설정해줍니다(아무데나 설정해도 오류는 안생깁니다. 원하는 폴더를 만드시거나 지정해주시면 됩니다.)- Next 누름

10. 각 ring 설정하기
- 한 sample의 염기서열은 하나의 ring이 됩니다. 두 개 이상도 하나의 ring 에 들어갈 수 있는 것 같아요..이 경우 나오는 그림을 어떻게 해석하는 건지는 모르겠네요.
- Ring 1:null 로 된 부분을 클릭하고 Data tool 에 있는 GC content를 클릭합니다. 첫 번째 ring은 sample의 염기서열이 아니라 reference sequence의 GC content를 보여줍니다. 이 걸 설정안하고 첫 번째 ring에 sample 염기서열을 넣어도 됩니다. 여러 번 해보면서 필요한 것만 선택해서 map을 그려보세요.
- 오른쪽 가운데 쯤 있는 "Add new ring"을 누르고 "Data pool" 에 있는 input 파일 줄 ring 2번에 넣고 싶은 input을 클릭한 후 "Add data"를 클릭합니다.
- 각 샘플당 하나 당 ring 하나씩 반복해서 만들면 됩니다.
- 뭔가 프로그램이 깔끔하지는 않아요. 더블클릭해야 해당 파일이 선택되기도 하고..아뭏든 불편하지만 안 되지는 않아요.
11. ring legend 만들기

"legent text:" 부분에 원하는 이름을 넣으시면 됩니다. Ring 1번은 GC content를 선택했었으니까 legent에는 GC content라고 적었습니다. Ring 2번부터 sample 의 염기서열을 넣었다면 ring 2번 부터의 legent는 각 sample의 이름을 넣어주면 됩니다.
Ring 색깔을 설정할 수도 있고 identity가 높은 것/낮은것 몇 %가 similar 할 때로 설정하겠는지 숫자로 넣어줄 수 있습니다.

Reference와 비슷한 염기서열을 보인다고 예상하시는데 좀 구분되는 지역을 보고싶다면 identity 설정을 높은 숫자로 넣어주시면 좀 더 명확하게 구분이 가능합니다. 지금 넣은 숫자들이 map을 그린 후 legend 부분에 반영되어 나옵니다. 숫자가 이상하다면 아마 similarity 설정이 통일되지 않아서 그럴거예요. Default 는 각각 70/ 50 입니다.
12. Circular map 그림 확인
Next를 누르고 그림 title 설정, Output file 위치 및 그림 파일 이름을 설정해줍니다. Submit 하고 잘 되면 아래와 같은 식으로 나홉니다.
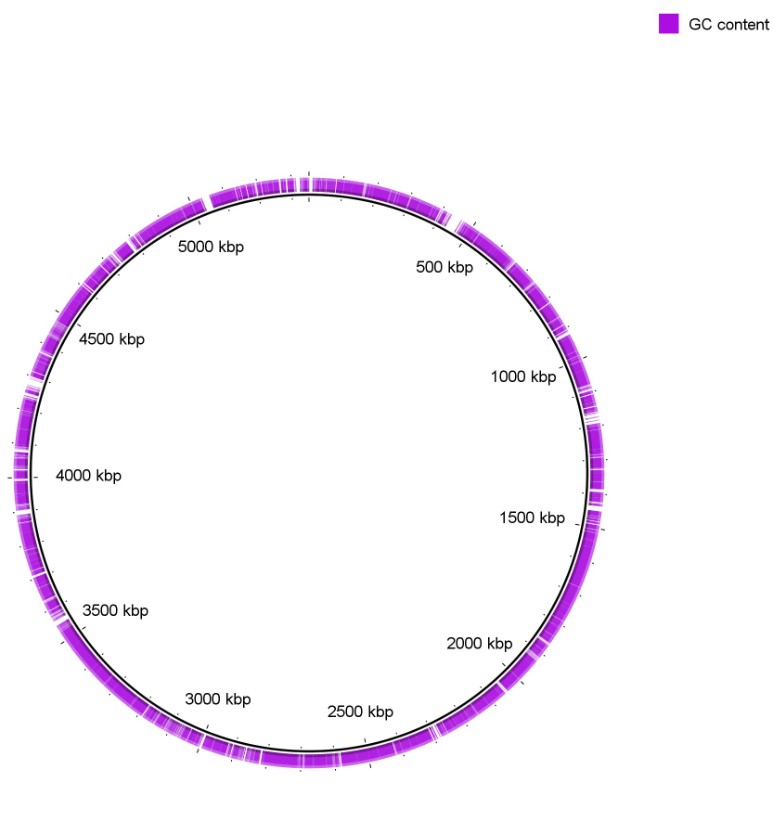
Ring legent 설정을 잘 못 했네요... 이렇게 나옵니다.... legend를 잘못 설정했네요 ㅠㅠㅠㅠ.
다시 잘 설정하면 아래와 같은 그림을 확인할 수 있습니다. 색깔이 에러네요

...* legend 부분때문에 circular map 부분이 잘리는 건 Preferences 부분의 image option에 size를 조절해주면 됩니다.
Gene Annotation, 원하는 부분 마킹도 가능합니다. 메뉴얼 읽어보면 어렵지 않아요. 그냥 수동으로 해도 되고, 엑셀 파일 형식으로 만든 후 넣어주면 자동으로 annotation 됩니다. 이건 다음에 채워놓을게요.
Circos (http://circos.ca/) 프로그램으로 만들어지는 그림이 훨씬 이쁜거 같지만...어떻게 사용하는지 모르겠네요. BRIG의 경우 설명이 긴 것 같지만 해보시면 어렵지 않아요. Circular map을 어떻게 그리는 지 모르신다면 BRIG으로 먼저 해보시고 다른 프로그램들을 돌려보는것도 좋을 것 같아요.
SMALL'genome data analysis > Genome analysis' 카테고리의 다른 글
Multiple genome alignment, Synteny map 그리기 - Mauve (0) 2020.04.18 Variants Call Format (VCF) 파일 하나로 합치기 (0) 2020.04.17 Local BLAST - makeblastdb 이용 nhr, nin, nsq 파일 만들기 (0) 2020.04.15 gbk 파일 변환하기 (0) 2020.04.11 Fastq to consensus sequences (1) 2020.04.04